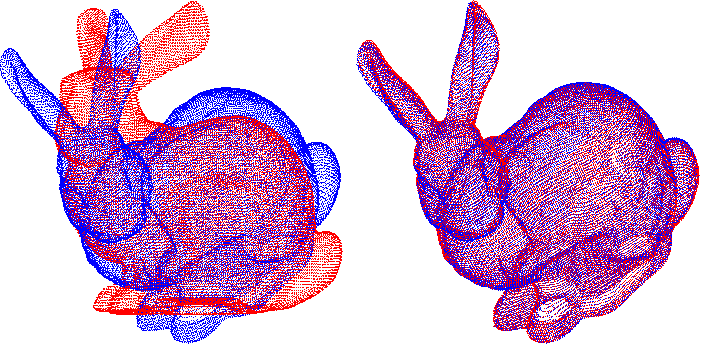
ICP (Iterative Closest Point) 와 Point Cloud Registration
2023, Apr 10
- 이번 글에서는 이론적으로
ICP (Iterative Closest Point)에 대한 내용과Cyrill Stachniss의ICP강의 내용을 정리해 보도록 하겠습니다.. - 강의는 총 1시간 분량의 강의 3개로 구성되어 총 3시간 분량의 강의입니다.
- 아래 참조 내용은 실제 코드 구현 시 도움 받은 내용입니다.
- 참조 : https://mr-waguwagu.tistory.com/36
- 참조 : https://github.com/minsu1206/3D/tree/main
목차
-
Matched Points의 Point-to-Point ICP 개념
-
Matched Points의 Point-to-Point ICP Python Code
-
Part 1: Known Data Association & SVD
-
Part 2: Unknown Data Association
-
Part 3: Non-linear Least Squares
Matched Points의 Point-to-Point ICP 개념
- 먼저 이번 글에서 다룰 강의 내용에 앞서서 간략하게 다룰 내용은 아래 강의 내용 중
Part 1: Known Data Association & SVD에 해당하는 내용입니다. 뒤의 강의 내용의 이해를 돕기 위하여 아래와 같이 먼저 정리하였습니다.
- 아래와 같이 2개의 점군 \(P, P'\) 가 있고 각 \(i\) 번째 점인 \(p_{i}, p'_{i}\) 가 서로 대응되는 것을 알고 있는 상황이라고 가정합니다.
- \[P = {p_{1}, p_{2}, ... , p_{n}}\]
- \[P' = {p'_{1}, p'_{2}, ..., p'_{n}}\]
- 위 점군에서 \(P\) 는
source에 해당하고 \(P'\) 는destination에 해당합니다. 즉, \(P \to P'\) 로 변환하기 위한 관계를 알고자 하는 것이 핵심입니다. - 따라서 \(P, P'\) 의 각 원소인 \(p_{i}, p'_{i}\) 의 관계를 알기 위해서는
Rotation을 위한 행렬 \(R\) 과Translation을 위한 벡터 \(t\) 가 필요합니다.
- \[\forall_{i} \ \ p'_{i} = Rp_{i} + t\]
- 이상적인 환경에서는 모든 \(i\) 에 대하여 \(p'_{i} = Rp_{i} + t\) 를 만족해야 하지만 현실적으로 오차가 포함되기 때문에 전체의 오차가 최소화 되는 방향으로 근사화 시키는 최적해를 구하는 방법을 이용하여
ICP를 적용합니다. RGB-D 카메라를 이용하거나 이미지에서Feature Extraction 및 Matching을 하여 점들끼리 쌍을 매칭한 점 군 \(P, P'\) 를 구한 경우에 지금부터 설명할 방법을 사용할 수 있습니다.
- 먼저 아래와 같이 \(i\) 점의 오차를 정의 합니다.
- \[e_{i} = p'_{i} - (Rp_{i} + t)\]
- 풀어야 할 문제는 모든 에러 \(e_{i}\) 를 최소화 시키는 목적 함수를 만들고 목적 함수를 최소화 시키는 문제를 푸는 것입니다. 따라서 다음과 같이
오차 제곱 합의 목적 함수를 만듭니다.
- \[\min_{R,t} \frac{1}{2} \sum_{i=1}^{n} \Vert (p'_{i} - (Rp_{i} + t)) \Vert^{2}\]
- 위 식에서 \(p_{i}, p'_{i}\) 는 벡터이기 때문에
norm을 적용하여 크기값인 스칼라 값으로 바꾸어서 목적 함수의 결과로 둡니다. - 위 식을 좀 더 단순화하여 전개하기 위해
두 점군의 중심 위치 (centroid)를 정의해 보도록 하겠습니다.
- \[p_{c} = \frac{1}{n} \sum_{i=1}^{n}(p_{i})\]
- \[p'_{c} = \frac{1}{n} \sum_{i=1}^{n}(p'_{i})\]
- 앞에서 정의한 목적 함수에 ① \(-p'_{c} + Rp_{c} + p'_{c} -Rp_{c} = 0\) 을 추가한 뒤 ②
제곱식을 전개해 보도록 하겠습니다.
- \[\begin{align} \frac{1}{2}\sum_{i=1}^{n} \Vert p'_{i} - (Rp_{i} + t) \Vert^{2} &= \frac{1}{2}\sum_{i=1}^{n} \Vert p'_{i} - Rp_{i} - t - p'_{c} + Rp_{c} + p'_{c} - Rp_{c} \Vert^{2} \\ &= \frac{1}{2}\sum_{i=1}^{n} \Vert (p'_{i} - p'_{c} - R(p_{i} - p_{c})) + (p'_{c} - Rp_{c} - t) \Vert^{2} \\ &= \frac{1}{2}\sum_{i=1}^{n} (\Vert p'_{i} - p'_{c} - R(p_{i} - p_{c}) \Vert^{2} + \Vert p'_{c} - Rp_{c} - t \Vert^{2} + 2(p'_{i} - p'_{c} - R(p_{i} - p_{c}))^{T}(p'_{c} - Rp_{c} - t)) \end{align}\]
- 위 식에서 다음 부분은 0이 됩니다.
- \[\sum_{i=1}^{n} (p'_{i} - p'_{c} - R(p_{i} - p_{c})) = 0\]
- 왜냐하면 모든 \(p'_{i}\) 의 총합과 \(p'_{c}\) 를 \(n\) 번 더한 것과 값이 같고 모든 \(p_{i}\) 의 총합과 \(p_{c}\) 를 \(n\) 번 더한 것과 값이 같기 때문입니다.
- 따라서 앞에서 전개한 식에서 \(\sum_{i=1}^{n} (p'_{i} - p'_{c} - R(p_{i} - p_{c}))\) 부분을 소거하면 다음과 같이 정리 가능합니다.
- \[\frac{1}{2}\sum_{i=1}^{n} (\Vert p'_{i} - p'_{c} - R(p_{i} - p_{c}) \Vert^{2} + \Vert p'_{c} - Rp_{c} - t \Vert^{2} + 2(p'_{i} - p'_{c} - R(p_{i} - p_{c}))^{T}(p'_{c} - Rp_{c} - t))\]
- \[\frac{1}{2}\sum_{i=1}^{n} (\Vert p'_{i} - p'_{c} - R(p_{i} - p_{c}) \Vert^{2} + \Vert p'_{c} - Rp_{c} - t \Vert^{2})\]
- \[\therefore \min_{R, t} J = \frac{1}{2}\sum_{i=1}^{n} ( \color{red}{\Vert p'_{i} - p'_{c} - R(p_{i} - p_{c}) \Vert^{2}} + \color{blue}{\Vert p'_{c} - Rp_{c} - t \Vert^{2}})\]
- 위 식의 빨간색 부분에 해당하는 항은
Rotation만 관련되어 있고 파란색 부분에 해당하는 항은Rotation과Translation모두 관련되어 있지만 추가적으로 \(p_{c}, p'_{c}\) 만 연관되어 있습니다. - 따라서 파란색 항은
Rotation만 구할 수 있으면 나머지 \(p_{c}, p'_{c}\) 는 주어진 점들을 통해 계산할 수 있으므로 \(\Vert p'_{c} - Rp_{c} - t \Vert^{2} = 0\) 으로 식을 두면 \(t\) 를 구할 수 있습니다.
- 빨간색 항 또한 조금 더 간단하게 만들기 위하여 다음과 같이 치환합니다.
- \[q_{i} = p_{i} - p_{c}\]
- \[q'_{i} = p'_{i} - p'_{c}\]
- 위 치환식의 정의에 따라서 \(q_{i}, q'_{i}\) 각각은 각 점 \(p_{i}, p'_{i}\) 가 점들의 중앙인 \(p_{c}, p'_{c}\) 로 부터 얼만큼 떨어져 있는 지 나타냅니다.
- 치환식을 이용하여 다음과 같은 2가지 스텝으로
Rotation과Translation을 구해보도록 하겠습니다.
- ①
Rotation\(R^{*}\) (예측값)를 다음 최적화 식을 통하여 구해보도록 하겠습니다.
- \[R^{*} = \text{argmin}_{R} \frac{1}{2} \sum_{i=1}^{n} \Vert q'_{i} - R q_{i} \Vert^{2}\]
- ② 앞에서 구한 \(R^{*}\) 을 이용하여 \(t^{*}\) 을 구합니다.
- \[t^{*} = p'_{c} - R^{*}p_{c}\]
- 먼저 ① 에 해당하는 \(R^{*}\) 을 구하는 방법에 대하여 살펴보도록 하겠습니다.
- \[\begin{align} \frac{1}{2} \sum_{i=1}^{n} \Vert q'_{i} - R q_{i} \Vert^{2} &= \frac{1}{2} \sum_{i=1}^{n}(q'_{i} - R q_{i} )^{T}(q'_{i} - R q_{i} ) \\ &= \frac{1}{2} \sum_{i=1}^{n}(q{'}_{i}^{t}q{'}_{i} + q_{i}^{t}R^{t}Rq_{i} - q{'}_{i}^{t}Rq_{i} - q_{i}^{t}R^{t}q'_{i}) \\ &=\frac{1}{2} \sum_{i=1}^{n}(q{'}_{i}^{t}q{'}_{i} + q_{i}^{t}q_{i} - q{'}_{i}^{t}Rq_{i} - (q_{i}^{t}R^{t}q'_{i})^{t}) \quad (\because q_{i}^{t}R^{t}q'_{i} \text{ : scalar} ) \\ &= \frac{1}{2} \sum_{i=1}^{n}(q{'}_{i}^{t}q{'}_{i} + q_{i}^{t}q_{i} -q{'}_{i}^{t}Rq_{i} -q{'}_{i}^{t}Rq_{i}) \\ &= \frac{1}{2} \sum_{i=1}^{n}(q{'}_{i}^{t}q{'}_{i} + q_{i}^{t}q_{i} -2q{'}_{i}^{t}Rq_{i}) \end{align}\]
- 위 식에서 첫번째 항과 두번째 항은 \(R\) 과 관련이 없습니다. 따라서 실제 최적화를 위한 함수는 다음과 같이 변경될 수 있습니다.
- \[\frac{1}{2} \sum_{i=1}^{n}(q{'}_{i}^{t}q{'}_{i} + q_{i}^{t}q_{i} -2q{'}_{i}^{t}Rq_{i}) \Rightarrow \frac{1}{2}\sum_{i=1}^{n} -2q{'}_{i}^{t}Rq_{i} = -\sum_{i=1}^{n}q{'}_{i}^{t}Rq_{i}\]
- 따라서 \(\min_{R, t} J\) 인 목적 함수를 최소화 하기 위해서는 \(\sum_{i=1}^{n}q{'}_{i}^{t}Rq_{i}\) 를
최대화하여 목적함수를 최소화 할 수 있도록 설계해야 합니다. 즉, \(\text{Maximize : } \sum_{i=1}^{n}q{'}_{i}^{t}Rq_{i}\) 를 만드는 것이 실제 풀어야할 최적화 문제가 됩니다.
- 그러면 \(\sum_{i=1}^{n}q{'}_{i}^{t}Rq_{i}\) 를 최대화 하기 위한 조건을 살펴보도록 하겠습니다.
- 식을 살펴보면 \(q_{i}, q'_{i}\) 는 벡터이고 \(R\) 은 3 x 3 크기의 행렬이므로 최종적으로 하나의 스칼라 값을 가지게 됩니다.
summation내부의 결과가 스칼라 값이므로trace연산( \(\text{Trace}()\) )의 성질을 이용할 수 있습니다.trace는 행렬의 대각 성분을 모두 더하는 연산입니다. 만약 최종 결과가 스칼라 값 (1 x 1 행렬)이고 이 값에trace연산을 적용하면 그 값 그대로 이기 때문에 값에 영향을 주지 않습니다. 따라서 결과값에 영향을 주지 않으면서trace연산의 성질들을 이용할 수 있습니다.trace연산의Cyclic Permutation성질은 다음을 만족합니다. 아래 기호 \(A, B, C\) 각각은 행렬입니다.
- \[\text{Trace}(ABC) = \text{Trace}(CAB) = \text{Trace}(BCA)\]
- 이 성질을 이용하여 앞에서 전개하였던 \(\sum_{i=1}^{n} q_{i}^{T} R q'_{i}\) 의 식을 변경해 보도록 하겠습니다.
- \[\begin{align} \sum_{i=1}^{n}q{'}_{i}^{t}Rq_{i} &= \text{Trace}\left(\sum_{i=1}^{n} q{'}_{i}^{t}Rq_{i} \right) \\ &= \text{Trace}\left(\sum_{i=1}^{n} q_{i}q{'}_{i}^{t}R \right) \\ &= \text{Trace}\left(\sum_{i=1}^{n} Rq_{i}q{'}_{i}^{t}\right) \\ &= \text{Trace}\left(R\sum_{i=1}^{n} q_{i}q{'}_{i}^{t}\right) \\ &= \text{Trace}(RH) \text{, where } \left(H = \sum_{i=1}^{n} q_{i}q{'}_{i}^{t}\right) \end{align}\]
- 위 식에서 \(q_{i} : (3 \times 1) \text{column vector}\) 이고 \(q{'}_{i}^{t} : (1 \times 3) \text{row vector}\) 이므로 \(\sum_{i=1}^{n} q_{i}q{'}_{i}^{t}\) 는 \(3 \times 3\) 행렬입니다. 따라서
SVD (Singular Value Decomposition)을 이용하여 행렬 분해를 할 수 있습니다. 특이값 분해 관련 내용은 아래 링크에 자세하게 설명되어 있습니다.특이값 분해: https://gaussian37.github.io/math-la-svd/
- 따라서 특이값 분해를 하면 다음과 같이 분해할 수 있습니다.
- \[H = \sum_{i=1}^{n} q'_{i} q_{i}^{T} = U \Sigma V^{T}\]
- 여기서 \(U, V\) 는
orthonormal matrix이고 \(\Sigma\) 는diagonal matrix이며 대각 성분은특이값을 가집니다.특이값과고유값은 다음의 관계를 가집니다.
- \[\text{singular value} = \sqrt{\text{eigen value}} > 0\]
SVD를 이용하여 분해한 값과 앞의 식을 이용하여 식을 좀 더 전개해 보도록 하겠습니다.
- \[\text{Trace}(RH) = \text{Trace}(R \sum_{i=1}^{n} q'_{i} q_{i}^{T}) = \text{Trace}(R U \Sigma V^{t})\]
- 위 식에서 최종적으로 구해야 하는 값은 \(R\) 이며 \(\text{Trace}(RH)\) 가 최대화 되도록 \(R\) 을 잘 설정하면 문제를 해결할 수 있습니다.
- 결론적으로 \(R = VU^{T}\)이 최적해의 솔루션이 됩니다. 최적해를 찾는 과정을 이해하기 위하여 다음 소정리(
Lemma)를 이용하도록 하겠습니다. - 소정리를 이해하기 위하여
Positive Difinite Matrix의 이해가 필요합니다. 관련 정의는 아래 링크에서 참조하시기 바랍니다.
- 먼저
Positive Definite Matrix를 만족하는 \(AA^{t}\) 행렬과orthonormal matrix\(B\) 가 있다고 가정하겠습니다. 이 때, 다음 식을 만족하는 것이 소정리 입니다.
- \[\text{Trace}(AA^{t}) \ge \text{Trace}(BAA^{t})\]
- 행렬 \(A\) 의 \(i\) 번째 열벡터를 \(a_{i}\) 로 가정하겠습니다. 이 때,
Trace의Cycle Permutation성질과Trace의 정의를 이용하면 다음과 같이 식을 전개할 수 있습니다.
- \[\begin{align} \text{Trace}(BAA^{t}) &= \text{Trace}(A^{t}BA) \\ &= \sum_{i}a_{i}^{t}(Ba_{i}) \end{align}\]
(Cauchy)Schwarz inequality를 이용하면 다음과 같이 식을 전개할 수 있습니다.(Cauchy)Schwarz inequality정의는 다음과 같습니다.
- \[\vert \langle u, v \rangle \vert \le \sqrt{\langle u, u \rangle} \cdot \sqrt{\langle v, v \rangle}\]
- \[u, v \text{ : vector}\]
- \[\langle u, v \rangle \text{ : inner product}\]
(Cauchy)Schwarz inequality의 정의에 따라 아래 식을 전개해 보도록 하겠습니다.
- \[a_{i}^{t}(Ba_{i}) \le \sqrt{(a_{i}^{t}a_{i})(a_{i}^{t}B^{t}Ba_{i})} = a_{i}^{t}a_{i} \quad (\because B \text{ : orthonormal matrix})\]
- \[\therefore \ \text{Trace}(BAA^{t}) \le \sum_{i} a_{i}^{t}a_{i} = \text{Trace}({AA^{t}})\]
- 위 성질을 이용하여 앞에서 증명하던 내용을 이어서 증명해 보도록 하겠습니다.
- \[H = U \Sigma V^{t}\]
- \[U, V \text{ : orthonormal matrix}\]
- \[\Sigma \text{ : diagonal matrix with singular value}\]
- \[\text{Let } X = VU^{t}\]
- 위 식에서 \(X\) 는
orthonormal matrix입니다. 왜냐하면 \(U, V\) 가 모두orthonormal matrix이기 때문에 그 곱또한orthonormal matrix를 만족하기 때문입니다.
- \[X = VU^{t}\]
- \[XH = VU^{t}U \Sigma V^{t} = V \Sigma V^{t}\]
Singular Value는 모두 0보다 크기 때문에eigenvalue는Singular Value의 제곱이므로 모든eigenvalue는 양수임을 알 수 있습니다.eigenvlaue가 모두 양수이면Positive Definite Matrix조건을 만족하기 때문에 (양의 정부호 행렬 (Positive Definite Matrix) 참조) \(XH\) 는Positive Definite Matrix입니다.- 따라서 앞에서 정리한 소정리를 이용할 수 있습니다.
- \[\text{Trace}(XH) \ge \text{Trace}(BXH) \quad (\forall \text{ orthonormal matrix } B )\]
- 이 내용을 앞에서 정리한 식과 연결해서 설명해 보도록 하겠습니다.
- \[\text{Trace}(RH) = \text{Trace}(VU^{T}H) \ge \text{Trace}(BRH)\]
- 즉, \(\text{Trace}(RH)\) 에서 \(R = VU^{T}\) 일 때, 모든 경우의 수에서
상한값을 가질 수 있으므로 최대화를 만족하기 위한 \(R^{*}\) 은 \(R^{*} = VU^{T}\) 을 통해서 구할 수 있습니다. - 이 때, 구한 \(R\) 의 \(\text{det}()\) (
determinant) 값은+1이어야 합니다. 왜냐하면 \(R\) 은orthonormal matrix이기 때문에determinant가+1을 만족하기 때문입니다. 하지만-1이 도출되는 경우가 발생할 수 있습니다. 이 경우는reflection이 발생한 경우이고 예외 케이스 입니다. 이 케이스를 처리하는 방법은 바로 뒤에서 살펴보도록 하겠습니다.
- 다음으로 ② 과정의 \(t^{*} = p'_{c} - R^{*}p_{c}\) 를 통해 간단하게 \(t^{*}\) 또한 구할 수 있습니다.
- 지금까지 살펴본 과정을 통해서 일반적인 상황에서의
Matched Points쌍에 대한Rotation과Translation을 구할 수 있었습니다. 그리고 도출 과정을 보면Rotation이 정상적으로 계산되면Translation은 부가적으로 얻을 수 있기 때문에Rotation을 잘 구하는 것이 핵심이 되는 것을 확인하였습니다. - 그러면
Rotation을 정상적으로 구할 수 있는 경우와 그렇지 않은 경우를 구분하여 어떤 케이스가 존재하는 지 살펴보도록 하겠습니다.
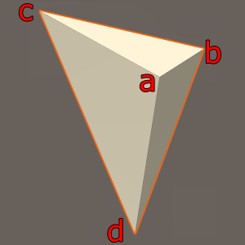
- ① \(q_{i}\) 가
coplanar가 아닌 경우 : 먼저 위 그림과 같이 점들이 하나의 평면에 있지 않는 경우가 가장 일반적인 상황입니다. 하나의 평면에 점들이 존재하는 상황을coplanar라고 하며 위 그림과 같은 상황은coplanar한 상황이 아닌 것으로 이해할 수 있습니다. 이 상황에서는Rotation의 솔루션이 유일하게 존재하므로SVD를 이용하여 구한 해를 정상적으로 사용할 수 있습니다.
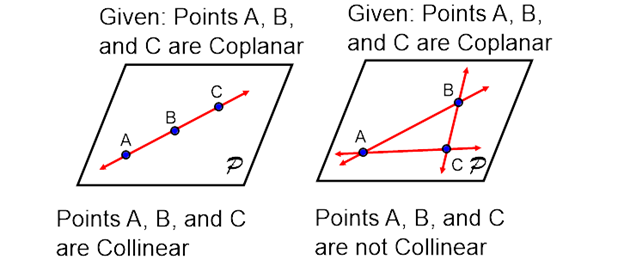
- ② \(q_{i}\) 가
colinear인 경우 : 두번째 케이스는 위 그림의 왼쪽과 같이 점들이coplanar이면서colinear인 경우 입니다. 이 경우에는 무한히 많은rotation의 경우의 수를 만들어 낼 수 있습니다. 따라서 이와 같은 경우에는 유일한 해를 구할 수 없습니다. 따라서 사전에 모든 점들이colinear인 지 확인해 보는 것이 중요합니다. 하지만 현실 데이터에서는 발생할 가능성이 매우 희박합니다.
- ③ \(q_{i}\) 가
coplanar이지만colinear가 아닌 경우 : 마지막 케이스는 위 그림에서 오른쪽에 해당합니다. 점들이coplanar이지만colinear가 아닌 경우입니다. 이 경우에도 ①과 마찬가지 방법으로 해를 구할 수 있습니다.
- 최종적으로 구한
Rotation이 정말Rotation으로써 의미를 가지려면orthonormal matrix이고determinant또한 +1을 가져야 합니다. - 하지만 경우에 따라서
determinant가 -1인 경우의 해를 얻을 수도 있습니다. 이와 같은 경우는Rotation이 아니라Reflection이 구해진 경우입니다. determinant가 -1인 경우 2가지로 해석할 수 있습니다.- ①
coplanar인 상태에서Reflection이 발생한 경우 \(V = [v_{1}, v_{2}, v_{3}] \to V = [v_{1}, v_{2}, -v_{3}]\) 로 변경하여Rotation을 구할 수 있습니다. 이 때, \(\lambda_{3} = 0\) 을 만족해야 합니다. - ②
determinant가 -1임에도 불구하고 \(\lambda_{3} \ne 0\) 인 경우에는 해를 구할 수 없습니다. 이 경우에는coplanar도 아니면서Rotation을 구하는 최적화를 하는 데 실패한 경우에 해당합니다. 이와 같은 상황은Noise가 많아서Rotation을 구하기 어려우므로Noise를 다루는RANSAC과 같은 방법을 사용하는 작업이 필요합니다.
- ①
- 그러면 바로 위의 ①에 해당하는 상황을 살펴보도록 하겠습니다.
coplanar에서는 모든 점들이 같은 평면에 있기 때문에, 차원이 하나 줄어든 것처럼 생각할 수 있습니다. 이 때,Rotation이 아닌Reflection이 발생하더라도 만족할 수 있습니다. - 모든 점들이
coplanar이면 앞에서 구한 \(H\) 의Singular Value하나가 0이 됩니다. 차원이 하나 소멸하기 때문에 발생한 문제이고 이와 같은 현상을degeneracy라고 합니다.
- \[H = \lambda_{1}u_{1}v'_{1} + \lambda_{2}u_{2}v'_{2} + 0 \cdot u_{3}v'_{3}\]
- \[u_{i}, v_{i} \text{ : column vector of } U, V\]
- \[\lambda_{1} > \lambda_{2} > \lambda_{3} = 0\]
- 따라서 위 케이스와 같이 행렬 분해가 된 경우 \(u_{3}\) 또는 \(v_{3}\) 의 부호를 바꾸더라도 \(H\) 에 영향을 주지 않습니다. 따라서 다음과 같이 \(V\) 를 변경합니다.
- \[V = [v_{1}, v_{2}, v_{3}] \to V' = [v_{1}, v_{2}, -v_{3}]\]
- 최종적으로 \(R = V'U^{T}\) 를 이용하여 \(R\) 을 구함으로써
Rotation을 정상적으로 구할 수 있습니다.
- 위 과정을 정리하면 다음과 같습니다.
- ① 행렬 \(H\) 를 구합니다.
- ② 행렬 \(H\) 의
SVD적용을 한 후 \(H = U \Sigma V^{T}\) 로 분해합니다. - ③ \(R = VU^{T}\) 를 이용하여 구합니다.
- ④ 만약 \(\text{det}(R) = 1\) 이면 정상적으로
Rotation이 구해진 상황입니다. - ⑤ 만약 \(\text{det}(R) = -1\) 이면 다음 2가지로 해석할 수 있습니다.
- ⑤-1 \(\lambda_{3} = 0\) 이면
coplanar이므로 \(R = V'U^{T}\) 로 구할 수 있습니다. - ⑤-2 \(\lambda_{3} \ne 0\) 이면 노이즈가 많아서 해를 구할 수 없는 상황입니다.
- ⑤-1 \(\lambda_{3} = 0\) 이면
Matched Points의 Point-to-Point ICP Python Code
- 지금부터 살펴볼 내용은 임의의
Rotation과Translation그리고 점군 \(P\) 를 생성한 다음 생성한Rotation과Translation을 이용하여 \(P' = R*P + t\) 를 통해 \(P'\) 를 만들어 보겠습니다. - 그 다음 \(P, P'\) 를 이용하여
ICP를 하였을 때, 생성한Rotation과Translation을 그대로 구할 수 있는 지 확인해 보도록 하겠습니다.
import numpy as np
from scipy.stats import special_ortho_group
def icp_svd(p_src, p_dst):
"""
Calculate the optimal rotation (R) and translation (t) that aligns
two sets of matched 3D points P and P_prime using Singular Value Decomposition (SVD).
Parameters:
- p_src: np.array of shape (3, n) -- the first set of points.
- p_dst: np.array of shape (3, n) -- the second set of points.
Returns:
- R: Rotation matrix
- t: Translation vector
"""
# Step 1: Calculate the centroids of P and P_prime
centroid_p_src = np.mean(p_src, axis=1, keepdims=True) # Centroid of P
centroid_p_dst = np.mean(p_dst, axis=1, keepdims=True) # Centroid of P'
# Step 2: Subtract centroids
q_src = p_src - centroid_p_src
q_dst = p_dst - centroid_p_dst
# Step 3: Construct the cross-covariance matrix H
H = q_src @ q_dst.T
# Step 4: Perform Singular Value Decomposition
U, Sigma, Vt = np.linalg.svd(H)
V = Vt.T
# Step 5: Calculate rotation matrix R
R_est = V @ U.T
result = True
# Step 6: Ensure R is a proper rotation matrix
det_R_est = np.linalg.det(R_est)
if np.abs(det_R_est - (-1)) < 0.0001: # check detminant
if np.abs(Sigma[-1]) < 0.00001 == 0: # check reflection
# Reflection in coplanar cse
V[:,-1] *= -1 # Flip the sign of the last column of V
R_est = V @ U.T
else:
# can't get rotation matrix
result = False
# Step 7: Calculate translation vector t
t_est = centroid_p_src - R_est @ centroid_p_dst
t_est = t.reshape(3, 1)
return R_est, t_est, result
# Example usage with dummy data
# Define the set of points P
P = np.random.rand(3, 30) * 100
# Set a random Rotation matrix R (ensuring it's a valid rotation matrix)
R = special_ortho_group.rvs(3)
# Set a random Translation vector t
t = np.random.rand(3, 1) * 10
# Apply the rotation and translation to P to create P_prime
P_prime = R @ P + t
################################### Calculate R and t using ICP with SVD
R_est, t_est, _ = icp_svd(P, P_prime)
print("R : \n", R)
print("R_est : \n", R_est)
print("R and R_est are same : ", np.allclose(R,R_est))
print("\n")
# R :
# [[-0.65800821 0.75067865 -0.05921784]
# [-0.56577368 -0.54475838 -0.61898179]
# [-0.49691583 -0.3737912 0.78316971]]
# R_est :
# [[-0.65800821 0.75067865 -0.05921784]
# [-0.56577368 -0.54475838 -0.61898179]
# [-0.49691583 -0.3737912 0.78316971]]
# R and R_est are same : True
print("t : \n", t)
print("t_est : \n", t_est)
print("t and t_est are same : ", np.allclose(t, t_est))
print("\n")
# t :
# [[7.19317157]
# [5.15828552]
# [2.92487954]]
# t_est :
# [[7.19317157]
# [5.15828552]
# [2.92487954]]
# t and t_est are same : True
- 위 코드의 결과와 같이 정상적으로 \(R, t\) 를 구할 수 있음을 확인하였습니다.
- 지금까지 살펴본 방법은 매칭이 주어질 때, \(R, t\) 를 추정하는 문제에 해당합니다.
- 매칭을 알고있는 경우에는 최소 제곱 문제를 해결하기 위한
analytic solution이 존재하기 때문에numerical solution을 이용한 최적화가 반드시 필요하진 않습니다. - 하지만 점들의 매칭에 오류가 있거나 점들의 \(X, Y, Z\) 값이 부정확한
outlier가 포함되면ICP를 진행하는 데 방해가 될 수 있습니다. 따라서 별도의outlier를 제거해야 좋은 \(R, t\) 값을 구할 수 있으므로outlier제거 알고리즘인RANSAC을 적용하여 정상적인 \(R, t\) 를 구하는 방법에 대하여 알아보도록 하겠습니다. RANSAC과 관련된 내용은 아래 링크를 참조하시기 바랍니다.RANSAC: https://gaussian37.github.io/vision-concept-ransac/
RANSAC을 이용할 때에는추출할 샘플 갯수,반복 시험 횟수,inlier threshold를 파라미터로 필요로 합니다. 그 부분은 추가적인 실험이나 위에서 공유한RANSAC개념 링크의 글을 통해 어떻게 파라미터를 셋팅하는 지 참조할 수 있습니다.- 아래 코드는 앞선 예제 코드에서
outlier데이터를 추가한 뒤RANSAC과정을 거쳐서 좀 더 강건하게Rotation과Translation을 구하는 예제입니다.
import numpy as np
from scipy.stats import special_ortho_group
def icp_svd(p_src, p_dst):
"""
Calculate the optimal rotation (R) and translation (t) that aligns
two sets of matched 3D points P and P_prime using Singular Value Decomposition (SVD).
Parameters:
- p_src: np.array of shape (3, n) -- the first set of points.
- p_dst: np.array of shape (3, n) -- the second set of points.
Returns:
- R: Rotation matrix
- t: Translation vector
"""
# Step 1: Calculate the centroids of P and P_prime
centroid_p_src = np.mean(p_src, axis=1, keepdims=True) # Centroid of P
centroid_p_dst = np.mean(p_dst, axis=1, keepdims=True) # Centroid of P'
# Step 2: Subtract centroids
q_src = p_src - centroid_p_src
q_dst = p_dst - centroid_p_dst
# Step 3: Construct the cross-covariance matrix H
H = q_src @ q_dst.T
# Step 4: Perform Singular Value Decomposition
U, Sigma, Vt = np.linalg.svd(H)
V = Vt.T
# Step 5: Calculate rotation matrix R
R_est = V @ U.T
result = True
# Step 6: Ensure R is a proper rotation matrix
det_R_est = np.linalg.det(R_est)
if np.abs(det_R_est - (-1)) < 0.0001: # check detminant
if np.abs(Sigma[-1]) < 0.00001 == 0: # check reflection
# Reflection in coplanar cse
V[:,-1] *= -1 # Flip the sign of the last column of V
R_est = V @ U.T
else:
# can't get rotation matrix
result = False
# Step 7: Calculate translation vector t
t_est = centroid_p_src - R_est @ centroid_p_dst
t_est = t.reshape(3, 1)
return R_est, t_est, result
def icp_svd_ransac(points_source, points_destination, n=3, num_iterations=20, inlier_threshold=0.01):
# n = 3 # Number of points to estimate the model, for affine 3D at least 4 points
# num_iterations = 20 # Number of iterations
# inlier_threshold = 0.1 # Inlier threshold, this might be a count or a percentage based on your needs
best_inliers = -1
best_R = None
best_t = None
for _ in range(num_iterations):
# Step 1: Randomly select a subset of matching points
indices = np.random.choice(points_source.shape[1], n, replace=False)
points_src_sample = points_source[:, indices]
points_dst_sample = points_destination[:, indices]
# Step 2: Estimate rotation and translation using SVD based ICP
R, t, result = icp_svd(points_src_sample, points_dst_sample)
# Step 3 and 4: Calculate error and inliers
points_src_transformed = R @ points_source + t
errors = np.linalg.norm(points_destination - points_src_transformed, axis=0)
inliers = np.sum(errors < inlier_threshold)
# Step 5: Check if current iteration has the best model
if inliers > best_inliers:
best_inliers = inliers
best_R = R
best_t = t
# Step 6: Check terminating condition
if best_inliers > inlier_threshold or _ == num_iterations - 1:
break
return best_R, best_t, best_inliers
# Example usage with dummy data
# Define the set of points P
P = np.random.rand(3, 30) * 100
# Set a random Rotation matrix R (ensuring it's a valid rotation matrix)
R = special_ortho_group.rvs(3)
# Set a random Translation vector t
t = np.random.rand(3, 1) * 10
# Apply the rotation and translation to P to create P_prime
P_prime = R @ P + t
# Add outliers to P_prime to create P_prime2
num_outliers = 10
P_prime2 = P_prime.copy()
P_prime2[:, -num_outliers:] = np.random.rand(3, num_outliers) * 100
################################## Calculate R and t using ICP with SVD, plus RANSAC
R_est, t_est, _ = icp_svd(P, P_prime2)
print("ICP without RANSAC : \n")
print("R : \n", R)
print("R_est : \n", R_est)
print("R and R_est are same : ", np.allclose(R,R_est))
print("\n")
# R :
# [[-0.65800821 0.75067865 -0.05921784]
# [-0.56577368 -0.54475838 -0.61898179]
# [-0.49691583 -0.3737912 0.78316971]]
# R_est :
# [[-0.33635851 0.94169333 -0.00875314]
# [-0.70013826 -0.25627327 -0.66643111]
# [-0.62981693 -0.21803137 0.74551523]]
# R and R_est are same : False
print("t : \n", t)
print("t_est : \n", t_est)
print("t and t_est are same : ", np.allclose(t, t_est))
print("\n")
# t :
# [[7.19317157]
# [5.15828552]
# [2.92487954]]
# t_est :
# [[7.19317157]
# [5.15828552]
# [2.92487954]]
# t and t_est are same : True
print("diff R and R_est : \n", np.linalg.norm(np.abs(R - R_est)))
print("\n")
# diff R and R_est :
# 0.5379242095232378
R_est, t_est, inliers = icp_svd_ransac(P, P_prime2)
print("ICP with RANSAC : \n")
print("R : \n", R)
print("R_est : \n", R_est)
print("R and R_est are same : ", np.allclose(R,R_est))
print("\n")
# R :
# [[-0.65800821 0.75067865 -0.05921784]
# [-0.56577368 -0.54475838 -0.61898179]
# [-0.49691583 -0.3737912 0.78316971]]
# R_est :
# [[-0.65800821 0.75067865 -0.05921784]
# [-0.56577368 -0.54475838 -0.61898179]
# [-0.49691583 -0.3737912 0.78316971]]
# R and R_est are same : True
print("t : \n", t)
print("t_est : \n", t_est)
print("t and t_est are same : ", np.allclose(t, t_est))
print("\n")
# t :
# [[7.19317157]
# [5.15828552]
# [2.92487954]]
# t_est :
# [[7.19317157]
# [5.15828552]
# [2.92487954]]
# t and t_est are same : True
print("diff R and R_est : \n", np.linalg.norm(np.abs(R - R_est)))
print("\n")
# diff R and R_est :
# 1.7603605962323948e-15
print("num inliers : ", inliers)
# num inliers : 20
icp_svd_ransac을 통하여outlier의 비율이 꽤 큰 경우에도 정상적인Rotation,Translation을 추정할 수 있음을 확인하였습니다.- 지금까지 살펴본 내용은 두 점군의 쌍을 매칭할 수 있을 때,
analytic solution을 이용하여 최적해를 구하는 방법에 대하여 알아보았습니다.
Part 1: Known Data Association & SVD
- 위 내용은 앞에서 다룬
Matched Points의 Point-to-Point ICP 개념과 일치합니다.