U-Net, Convolutional Networks for Biom edical Image Segmentation
2019, Oct 02
- 이번 글에서는 U-Net에 대하여 알아보도록 하겠습니다.
목차
-
U-Net 이란
-
Overlap-tile
-
U-Net의 전체 구조
-
pytorch 코드
U-Net 이란

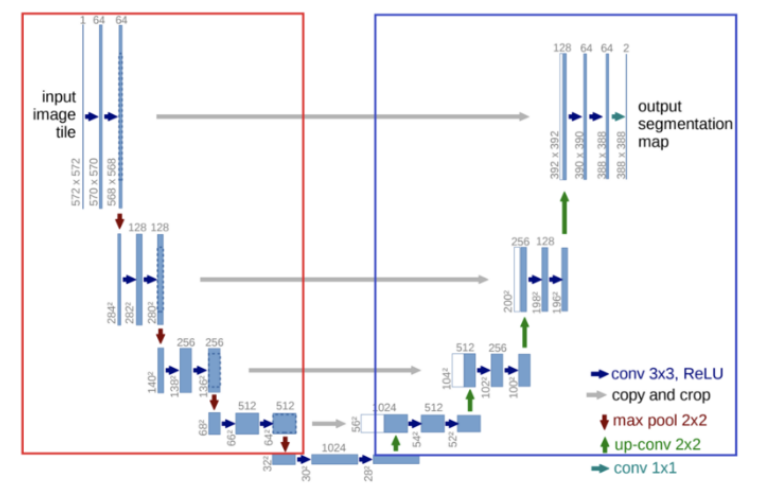
U-net은 2015년에 위 그림과 같이 U 커브 형태의 Convolutional Layer를 쌓은 구조로Segmentaion을 하기 위한 네트워크 구조입니다.- 이 모델은 생체 데이터 이미지에서 세그멘테이션 작업을 수행하기 위해 만들어 졌습니다.
- 예를 들어 세포 단면을 찍은 사진에서 세포 내부에서 구분하고 싶은 부분들의 위치를 분할하기 위해서 사용된 것입니다.
- 이 구조는 크게 2가지로 나뉘어져 있는데 첫번째로 빨간색 박스에 해당하는
downsampling과정과 파란색 박스에 해당하는upsampling과정이 있습니다. - downsampling 과정을 논문에서는
Constracting Path라고 하였는 데, 이 path에서의 역할은 이미지의Context를 보는 것입니다.- 즉, 이미지에서 전체적인 구성을 보는 것입니다. 이미지에서 어떤 물체가 있는지에 대한 정보등을 얻게 됩니다.
- 반면 upsampling 과정은
Expanding Path라고 하고, 이 path에서의 역할은Localization입니다. 즉, 물체의 위치를 찾는 것입니다. - 전체적으로 보면
constracting path에서는 pooling을 통하여 resolution을 줄여 나아가고expanding path에서는 다시 resolution을 높여 나아가는 구조입니다.
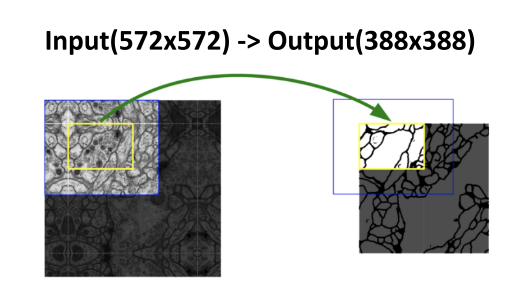
U-net에서의 입출력을 보려면 위 이미지의 좌(입력) 우(출력)을 살펴보면 됩니다. 입력은 세포 단면의 이미지이고 출력은 분할된 이미지가 나오게 됩니다. 화살표 시작점은 이미지가 복잡한데 화살표 끝점의 이미지는 간단해 보이는게 세포 내부/외부의 분할된 결과를 표시하기 때문입니다.- 좀 더 자세하게 말하면 입력 이미지는 채널이 하나인 흑백 이미지이고 모델의 결과로 나오는 텐서는 채널이 2개인 텐서입니다.
- 각각의 채널은 해당 픽셀이 세포의 경계선에 해당하는지 아니면 세포의 경계선이 아닌지에 대한 수치라고 볼 수 있습니다.
- 예를 들어 결과값의 크기가 256 x 256이고 특정 값인 (10, 10)을 살펴보면 2개의 채널로 값을 가지고 있습니다.
- 그값이 [0.5, -0.1]이라면 소프트맥스 연산을 통해 총합이 1인 확률 값으로 만들 수 있고 높은 확률값에 해당하는 클래스를 선택하면 됩니다.
- 첫번째 클래스가 세포의 경계선, 두번째 클래스가 세포의 내부라고 한다면 위의 결과는 세포의 경계선이 될 것입니다.
Overlap-tile
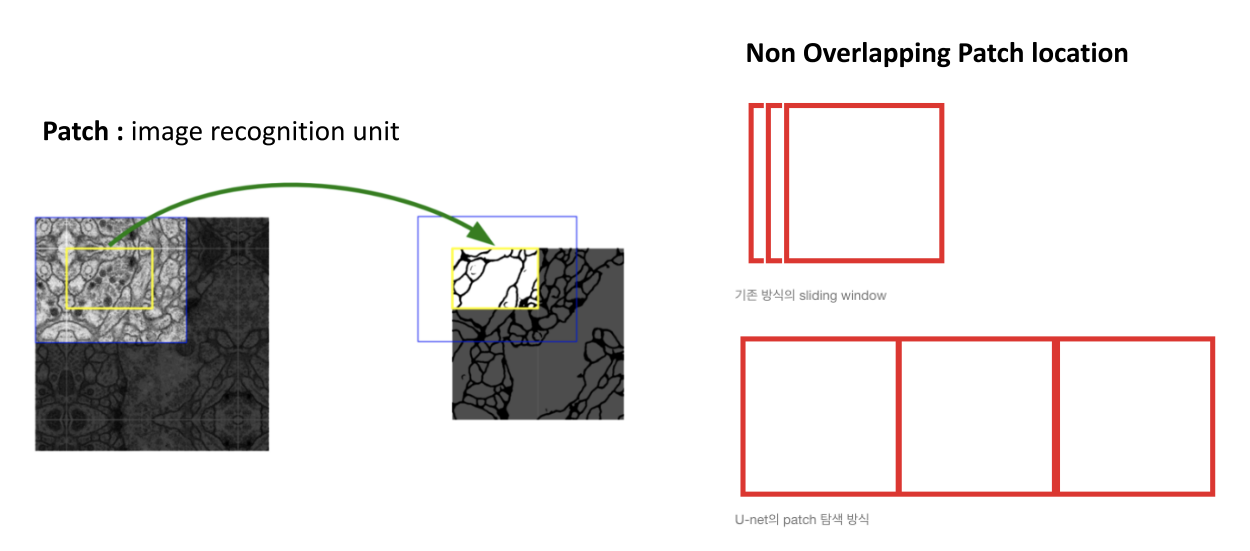
U-net에서 사용한 image recognition의 기본 단위는patch입니다.- 기존의 슬라이딩 윈도우 방식에서는 오른쪽 그림과 같이 patch가 겹쳐진 형태로 탐색이 되었지만 U-net에서는 segmentation 작업인 만큼 속도에 문제가 있기 때문에 속도 향상을 위하여 오버랩 되는 구간 없이 Patch를 탐색합니다.
- 즉, Patch를 안겹치도록 배치하고 탐색하여 넓은 영역을 빠른 속도로 살펴 보자는 것이 컨셉입니다.
- 물론 지금 다루고 있는 것이
Overlap-tile인 것 처럼 바로 아래에서 어떻게 overlap이 되는 것인지 알아보겠습니다.
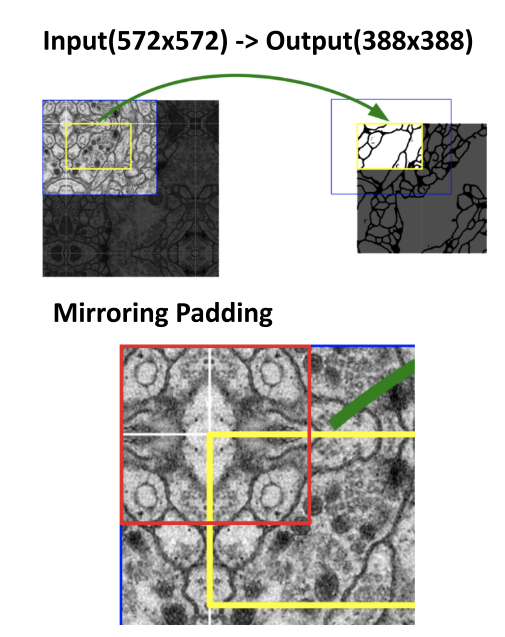
- patch를 퍼즐 맞추듯이 전체 이미지에서 타이트 하게 배치 시키면 전체 사이즈와 맞지 않아 패딩이 필요하게 됩니다.
- U-net에서는 일반적으로 많이 사용하는 zero 패딩이 아닌
Mirroring padding이 사용 되었습니다. - 즉, patch 기준으로 패딩 해야 하는 부분을 거울에 비치듯 대칭이 되도록 복사하고 그 값으로 패딩 영역을 채워넣은 것입니다. 위 그림을 보면 쉽게 이해할 수 있습니다.
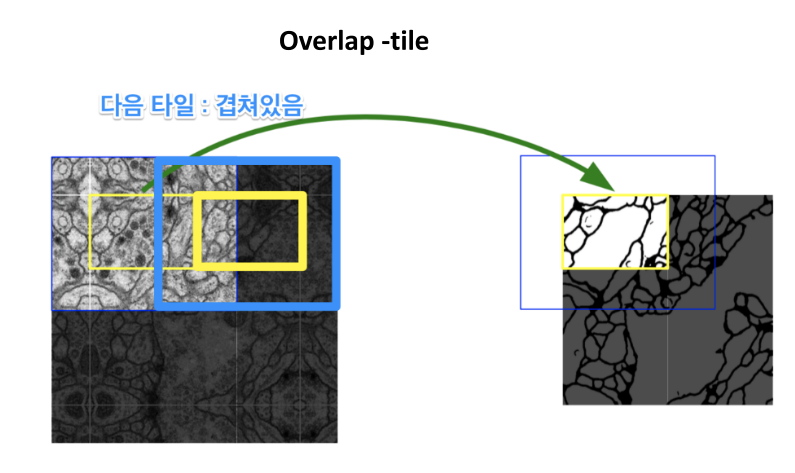
- 그러면 patch에 mirroring padding을 적용하게 되면 patch + mirroring padding 간에는 겹치는 구간이 발생하게 됩니다.
- 이 단위를
tile이라고 하고 tile 끼리는 겹치기 때문에overlap-tile이라는 이름으로 소개되었습니다. - 일반적으로 mirroring padding을 잘 사용하지는 않는데, u-net은 의학용 데이터를 기반의 논문이었고 의학용 데이터 영역에서는 의미가 있기 때문에 사용하지 않았나 추측이 됩니다.
- 따라서 이후의 pytorch 코드에서는 범용적으로 사용하는 zero padding으로 구현을 하려고 하오니 참조하시기 바랍니다.
U-Net의 전체 구조
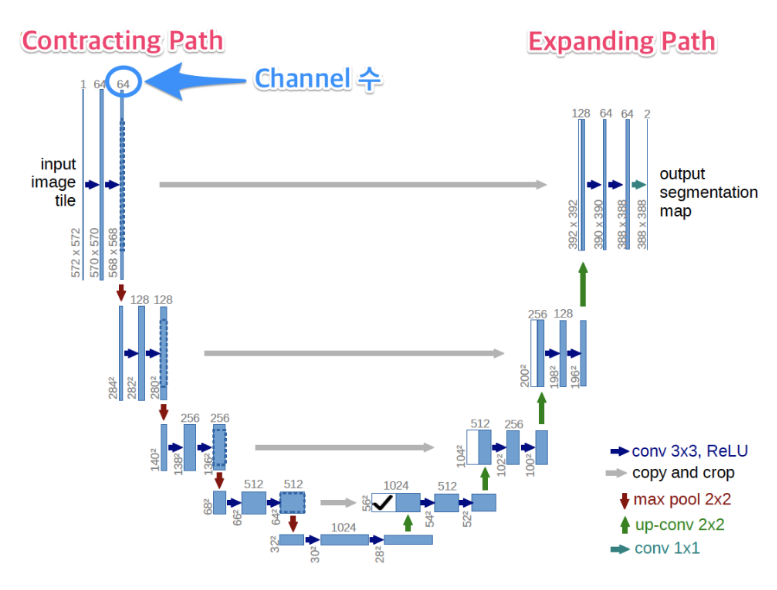
- 앞에서 설명한 바와 같이 U-net은 크게 constracting path와 expanding path로 이루어져 있습니다.
- 먼저 constracting path의 구조를 살펴보겠습니다. 여기에서는 크게 다음 요소들로 구성되어 있습니다.
- 3x3 convolution + ReLU
- 2x2 max pooling stride 2
- downsample 시 feature의 크기는 1/2배, channel의 수는 2배
- 다음으로 expanding path의 구조를 살펴보겠습니다.
- 3x3 convolution + ReLU
- upsample 시 feature의 크기는 2배(2x2 up-convolution), channler의 수는 1/2배
- copy and crop : contracting path의 feautre를 copy한 다음 그림에서와 같이 expanding path의 대칭되는 계층에
concatenation을 합니다. 이 때 contracting path와 expanding path의 feature 크기가 다르므로 contracting path의 feature를 copy한 다음 concatenation을 할 expanding path의 feature 사이즈에 맞추어서 crop을 합니다. 따라서 이 작업을copy and crop이라고 합니다.
- 위에서 말씀드린 바와 같이 U-net에서의 skip-connection의 방식은
concatenation입니다. sum이 아니지요. - backbone network와 비교해 본다면,
ResNet에서의 skip-connection은 sum 입니다. 즉 실제 element-wise로 덧셈 연산이 이루어 집니다. - 반면
DenseNet에서는 skip-connection이 sum이 아니라 channel 방향으로 concatenation 하는 것입니다. - segmentation과 비교해 보면 FCN은 skip-connection 사용 시 ResNet과 유사하게 sum 연산을 이용하는 반면
U-net은 DenseNet과 유사하게 concatenation 연산을 한다고 생각하시면 됩니다.
U-net을Encoder-Decoder관점에서 살펴보겠습니다.Encoder-Decoder방식의 대표적인 네트워크가 오토인코더 입니다.- 오토인코더에서는 입력 이미지가 압축되다 보면 위치 정보가 어느 정도 손실되게 됩니다.
- 그렇게 되면 다시 원본 이미지 크기로 복원하는 과정 속에서 정보의 부족 때문에 원래 물체가 있었던 위치에서 어느 정도 이동이 발생하게 됩니다.
- 이런 복원 과정에 skip connection을 사용하게 되면 원본 이미지의
위치 정보를 추가적으로 전달받는 셈이 되므로 비교적으로 정확한 위치를 복원할 수 있게 되고 segmentation 결과도 좋아지게 됩니다.
pytorch 코드
- 코드 전체 : https://github.com/gaussian37/Deep-Learning-Implementation/blob/master/u-net/u_net.py
- 논문에서는 세포의 이미지를 흑백으로 구분하기 때문에 입력 채널이 1이지만 일반적인 경우 컬러 이미지를 사용하기 때문에 3개의 채널이 들어오는 형태가 되도록 하면 좀 더 general 하게 사용할 수 있습니다.
- 즉 컬러 이미지가 들어오고 K개의 클래스로 나뉜다면 입력 채널은 3, 출력 채널은 K가 되어야 합니다.
U-net이나 다른 segmentation 모델을 보면 반복되는 구간이 꽤 많기 때문에block에 해당하는 클래스를 만들어 사용하면 편하게 구현할 수 있습니다.- 아래 코드는 convolution layer, batch normalization, activation function을 묶어서 convolution block을 만든 것입니다.
def ConvBlock(in_dim, out_dim, act_fn):
model = nn.Sequential(
nn.Conv2d(in_dim, out_dim, kernel_size = 3, stride = 1, padding = 1),
nn.BatchNorm2d(out_dim),
act_fn,
)
return model
def ConvTransBlock(in_dim, out_dim, act_fn):
model = nn.Sequential(
nn.ConvTranspose2d(in_dim, out_dim, kernel_size = 3, stride = 2, padding=1, output_padding = 1),
nn.BatchNorm2d(out_dim),
act_fn,
)
return model
def Maxpool():
pool = nn.MaxPool2d(kernel_size = 2, stride = 2, padding = 0)
return pool
def ConvBlock2X(in_dim, out_dim, act_fn):
model = nn.Sequential(
ConvBlock(in_dim, out_dim, act_fn),
ConvBlock(out_dim, out_dim, act_fn),
)
return model
- 이 블록들을 사용해서 U-net 모델의 클래스를 작성해 보겠습니다.
- 아래 코드에서
in_dim은 입력되는 채널의 수,out_dim은 최종 결과값의 채널 수(또는 클래스 수),num_filter는 convolution 연산 중간 중간에 사용할 필터의 수 기준값입니다.
class UNet(nn.Module):
def __init__(self, in_dim, out_dim, num_filter):
super(UNet, self).__init__()
self.in_dim = in_dim
self.out_dim = out_dim
self.num_filter = num_filter
act_fn = nn.LeakyReLU(0.2, inplace = True)
self.down_1 = ConvBlock2X(self.in_dim, self.num_filter, act_fn)
self.pool_1 = Maxpool()
self.down_2 = ConvBlock2X(self.num_filter, self.num_filter * 2, act_fn)
self.pool_2 = Maxpool()
self.down_3 = ConvBlock2X(self.num_filter * 2, self.num_filter * 4, act_fn)
self.pool_3 = Maxpool()
self.down_4 = ConvBlock2X(self.num_filter * 4, self.num_filter * 8, act_fn)
self.pool_4 = Maxpool()
self.bridge = ConvBlock2X(self.num_filter * 8, self.num_filter * 16, act_fn)
self.trans_1 = ConvTransBlock(self.num_filter * 16,self.num_filter * 8, act_fn)
self.up_1 = ConvBlock2X(self.num * 16, self.num_filter * 8, act_fn)
self.trans_2 = ConvTransBlock(self.num_filter * 8, self.num_filter * 4, act_fn)
self.up_2 = ConvBlock2X(self.num_filter * 8, self.num_filter * 4, act_fn)
self.trans_3 = ConvTransBlock(self.num_filter * 4, self.num_filter * 2, act_fn)
self.up_3 = ConvBlock2X(self.num_filter * 2, self.num_filter, act_fn)
self.trans_4 = ConvTransBlock(self.num_filter * 2, self.num_filter, act_fn)
self.up_4 = ConvBlock2X(self.num_filter *2, self.num_filter, act_fn)
self.out = nn.Sequential(
nn.Conv2d(self.num_filter, self.out_dim, 3, 1, 1),
nn.LeakyReLU(0.2, inplace = True),
)
def forward(self, input):
down_1 = self.down_1(input) # concat w/ trans_4
pool_1 = self.pool_1(down_1)
down_2 = self.down_2(pool_1) # concat w/ trans_3
pool_2 = self.pool_2(down_2)
down_3 = self.down_3(pool_2) # concat w/ trans_2
pool_3 = self.pool_3(down_3)
down_4 = self.down_4(pool_3) # concat w/ trans_1
pool_4 = self.pool_4(down_4)
bridge = self.bridge(pool_4)
trans_1 = self.trans_1(bridge)
concat_1 = torch.cat([trans_1, down_4], dim = 1)
up_1 = self.up_1(concat_1)
trans_2 = self.trans_1(up_1)
concat_2 = torch.cat([trans_2, down_3], dim = 1)
up_2 = self.up_1(concat_2)
trans_3 = self.trans_1(up_2)
concat_3 = torch.cat([trans_3, down_2], dim = 1)
up_3 = self.up_1(concat_3)
trans_4 = self.trans_1(up_3)
concat_4 = torch.cat([trans_4, down_1], dim = 1)
up_4 = self.up_1(concat_4)
out = self.out(up_4)
return out
- forward 함수를 보면
Encoder부분에서는 주어진 연산을 계속 하고Decoder부분에서는torch.cat을 통하여concatenation연산이 발생하는 것을 확인할 수 있습니다.